Datenexploration
Dies ist ein Tutorial zum Selbststudium von Johannes Gerwien.
- Version 1.0.0, April 2022
- gefundene Fehler und Feedback bitte an gerwien@idf.uni-heidelberg.de
1 Ziel
Dieses kurze Manual soll dabei helfen, Daten auf einfache Weise zu erkunden (“Datenexploration”). Es setzt voraus, dass Nutzer bereits eine ungefähre Vorstellung von der Arbeit mit R bzw. R Studio haben. Ein R manual für Anfänger finden Sie hier.
Es handelt sich bei diesem Manual eigentlich um nichts weiter als eine Zusammenstellung von dem Code, den ich in fast allen meinen Projekten verwende. Dabei liegt der Fokus vor allem auf Einfachheit.
Wir beschäftigen uns mit zwei Ansätzen:
Insbesondere die Datenexploration mit Grafiken hilft fast immer dabei, die Daten in einem ersten Schritt zu “verstehen” und/oder Fehler bei der Kodierung zu erkennen. Außerdem sind die Mittel für die Datenexploration, die ich hier zeige, so einfach, dass sie sich auch eignen, um Zwischenergebnisse zu betrachten - also während die Datenerhebung noch läuft.
2 Vorbereitung
2.1 Lade Pakete (“libraries”)
- der folgende Code überprüft, ob alle für dieses Manual notwendigen Pakete installiert sind; wenn nicht werden sie heruntergeladen und installiert
requiredPackages <- c("ggplot2",
"janitor",
"Hmisc",
"plyr",
"scales")
install_pack <- function(pkg){
new.pkg <- pkg[!(pkg %in% installed.packages()[, "Package"])]
if (length(new.pkg))
install.packages(new.pkg, dependencies = TRUE)
sapply(pkg, require, character.only = TRUE)
}
install_pack(requiredPackages)## ggplot2 janitor Hmisc plyr scales
## TRUE TRUE TRUE TRUE TRUE2.2 Daten laden
- Wir laden einen Testdatensatz: Download
- … und speichern ihn im Projektordner ( siehe R-Manual für Anfänger zum Thema “Stay organized” )
- wir laden den Datensatz mit der Funktion read.csv()
- alternativ kann man den Datensatz natürlich auch über die Import-Funktion in R studio laden
data <- read.csv("test_set3.csv", stringsAsFactors = TRUE)2.3 Der Datensatz
- wir schauen uns an, was im Datensatz enthalten ist
str(data)## 'data.frame': 1582 obs. of 7 variables:
## $ subject : int 1 1 1 1 1 1 1 1 1 1 ...
## $ condition1 : Factor w/ 2 levels "AA","AI": 2 2 2 2 1 2 2 1 1 1 ...
## $ condition2 : Factor w/ 2 levels "agent","patient": 1 1 1 1 1 1 1 1 1 1 ...
## $ item : int 38 9 31 4 1 23 25 32 22 15 ...
## $ trial_ID : int 36 27 9 46 35 6 75 76 61 63 ...
## $ dep_V_numeric : int 1520 1360 1673 1383 2140 1848 1730 1925 1053 1967 ...
## $ dep_var_binary: int 1 1 1 1 1 1 1 1 1 1 ...- der Datensatz simuliert Daten, wie sie typischerweise in psychologischen oder psycholinguistischen Experimenten vorkommen
- er enthält 7 Spalten:
- subject - eine eindeutige ID für jede Versuchsperson
- condition1 - eine Variable mit zwei Ausprägungen (unabhängige Variable 1)
- condition2 - eine zweite Variable mit zwei Ausprägungen (unabhängige Variable 2)
- item - eine eindeutige ID für jedes Item
- trial_ID - eine eindeutige ID für den Versuchsdurchgang
- dep_V_numeric - eine numerische Variable; sie stellt die Reaktion einer Versuchsperson in einem Trial dar und ist damit eine abhängige Variable; in diesem Fall Reaktionszeiten (reaction time = “RT”)
- dep_var_binary - eine weitere abhängige Variable; in diesem Fall eine Variable, die das Auftreten einer bestimmten Reaktion erfasst (ja=1, nein=0); eine binäre Variable
3 Datenexploration in Zahlen
3.1 Frequenz
- Frequenz bedeutet Häufigkeit eines bestimmten Merkmals (Ausprägung einer Variable) - in vielen Fällen, Häufigkeit einer bestimmten Reaktion durch Versuchsteilnehmende, zum Beispiel:
- ja-/nein-Antworten
- Verwendung einer bestimmten syntaktischen Struktur
- Auftreten eines Fehlers
- …
- ich verwende hier das Paket “Janitor”; das lädt man ganz normal mit library(janitor)
3.1.1 ganzer Datensatz
- die Variable “dep_var_binary” in unserem Testdatensatz kodiert, ob eine Reaktion auftritt bzw. nicht auftritt (1/0)
- wir überprüfen zuerst, in wieviel Prozent eine “1” bzw. eine “0” in unseren Daten auftritt
library(janitor)
tabyl(data, dep_var_binary)%>%
adorn_percentages("col") %>%
adorn_pct_formatting(digits = 1)## dep_var_binary n percent valid_percent
## 0 0.4 44.2% 45.2%
## 1 0.5 53.5% 54.8%
## NA 0.0 2.2% -- eine 0 tritt in 44.2% auf, eine 1 in 53.5% und keine Angabe (“NA”) in 2.2%
- schließt man NA-Werte aus, tritt in 45.2% eine 0 und in 54.8% eine 1 auf
- wir können uns das Ganze auch als Zahl - nicht in Prozent - ausgeben lassen
tabyl(data, dep_var_binary)## dep_var_binary n percent valid_percent
## 0 700 0.44247788 0.4524887
## 1 847 0.53539823 0.5475113
## NA 35 0.02212389 NA- in 700 Datenzeilen gibt es eine 0, in 847 eine 1 und in 35 eine NA-Angabe
3.1.2 eine Bedingung
- wir haben zwei Variablen (condition1, condition2) mit jeweils zwei Ausprägungen:
- condition1: AA, AI
- condition2: agent, patient
- wie häufig sind 1 und 0 in condition1?
tabyl(data, dep_var_binary, condition1)%>%
adorn_percentages("col") %>%
adorn_pct_formatting(digits = 1)## dep_var_binary AA AI
## 0 42.0% 46.5%
## 1 55.4% 51.7%
## NA 2.7% 1.8%- wie häufig sind 1 und 0 in condition2?
tabyl(data, dep_var_binary, condition2)%>%
adorn_percentages("col") %>%
adorn_pct_formatting(digits = 1)## dep_var_binary agent patient
## 0 7.2% 81.3%
## 1 90.0% 17.1%
## NA 2.8% 1.6%3.1.3 mehrere Bedingungen
- wir können auch mehrere Bedingungen kombinieren, hier condition1 und condition2
tabyl(data, dep_var_binary, condition1, condition2)%>%
adorn_percentages("col") %>%
adorn_pct_formatting(digits = 1)## $agent
## dep_var_binary AA AI
## 0 9.6% 4.8%
## 1 86.9% 93.1%
## NA 3.5% 2.0%
##
## $patient
## dep_var_binary AA AI
## 0 74.6% 87.9%
## 1 23.6% 10.6%
## NA 1.8% 1.5%3.2 Mittelwerte und Varianz
der Mittelwert gibt den Durschnitt über mehrere Messungen an
Man kann für sehr viele Dinge den Durchschnitt berechnen, zum Beispiel:
- durchschnittliches Auftreten einer Antwort
- durchschnittle Präferenz für A gegenüber B
- Reaktionszeiten
- …
Der Mittelwert selbst ist aber nicht besonders aussagekräftig, wenn man nicht auch die Varianz kennt, also den Bereich, in dem alle oder die meisten Messwerte liegen
wir berechnen hier als Maß für die Varianz zusätzlich zum Mittelwert die Standardabweichung
Unterschied zwischen Standardabweichung (SD) und Standardfehler (SE): hier auf youtube
wir benutzen hier ddply aus dem Paket plyr; man lädt plyr ganz normal mit library(plyr)
mehr zu Zusammenfassungen mit “ddply” cookbook for R
3.2.1 gesamter Datensatz
- unser Testdatensatz enthält eine numerische Variable, nämlich “dep_V_numeric”
- wir betrachten diese Werte als Beispiele für Reaktionszeiten
- wir berechnen zuerst Mittelwerte und Standardabweichung für den gesamten Datensatz
library(plyr)
ddply(data, .(), summarize,
Mittelwert = mean(dep_V_numeric, na.rm=T),
Standardabweichung = sd(dep_V_numeric, na.rm = T))## .id Mittelwert Standardabweichung
## 1 <NA> 1970.614 783.07473.2.2 eine Bedingung
- Mittelwerte und Standardabweichung für condition1
ddply(data, .(condition1), summarize,
Mittelwert = mean(dep_V_numeric, na.rm=T),
Standardabweichung = sd(dep_V_numeric, na.rm = T))## condition1 Mittelwert Standardabweichung
## 1 AA 1991.589 784.4395
## 2 AI 1949.638 781.64043.2.3 mehrere Bedingungen
- Mittelwerte und Standardabweichung für condition1 und condition2
ddply(data, .(condition1, condition2), summarize,
Mittelwert = mean(dep_V_numeric, na.rm=T),
Standardabweichung = sd(dep_V_numeric, na.rm = T))## condition1 condition2 Mittelwert Standardabweichung
## 1 AA agent 1977.879 811.6258
## 2 AA patient 2005.404 756.8406
## 3 AI agent 1909.297 760.8597
## 4 AI patient 1989.675 800.68073.2.4 weitere Anwendungen
- Mittelwerte pro Versuchsperson für condition1 und condition2
- dieses Mal speichern wir das Ergebnis in einem neuen Objekt; hier “by_sub”
by_sub <- ddply(data, .(subject, condition1, condition2), summarize,
Mittelwert = mean(dep_V_numeric, na.rm=T),
Standardabweichung = sd(dep_V_numeric, na.rm = T))- … und lassen uns anschließend die Ergebisse für die ersten 10 Zeilen ausgeben
- Weil jede Versuchsperson Daten für alle 4 experimentellen Bedingungen produziert hat (condition1 mit 2 Ausprägungen, condition2 mit zwei Ausprägungen), gibt es für jede Versuchperson genau vier Zeilen mit Mittelwerten und Standardabweichungen
head(by_sub, 10)## subject condition1 condition2 Mittelwert Standardabweichung
## 1 1 AA agent 1764.4 478.9545
## 2 1 AA patient 1566.8 521.1839
## 3 1 AI agent 1598.7 248.6055
## 4 1 AI patient 1804.1 453.0127
## 5 2 AA agent 1656.0 263.5130
## 6 2 AA patient 1923.7 421.5316
## 7 2 AI agent 1825.7 235.4136
## 8 2 AI patient 1666.7 274.4433
## 9 3 AA agent 1443.4 351.1325
## 10 3 AA patient 1656.2 286.50574 Visuelle Datenexploration
- für die Grafiken benötigen wir das package ggplot2; man lädt ggplot2 ganz normal mit library(ggplot2)
- Fast alles zu Grafiken mit ggplot2 gibt es auf http://www.cookbook-r.com/Graphs/
4.1 Frequenz (Häufigkeit)
- für den Ansatz, den ich hier für die Darstellung von Häufigkeiten beschreibe, muss die abhängige Variable als Faktor kodiert werden
data$dep_var_binary <- factor(data$dep_var_binary, levels = c("1","0"))4.1.1 eine Variable
- facet_grid() erlaubt uns die Darstellung in Panels (Infos zu “facet_grid()” )
- Prozentangaben werden automatisch berechnet
library(ggplot2)
ggplot(data=data, aes(x= dep_var_binary, group=condition1)) +
geom_bar(aes(y = ..prop.., fill = factor(..x..)), stat="count") +
geom_text(aes( label = scales::percent(..prop.., accuracy = 0.1),
y= ..prop.. ), stat= "count", vjust = -.5, size = 2.5) +
labs(y = "percent", fill="AV") +
facet_grid(~ condition1) +
scale_y_continuous(labels = scales::percent)+
scale_x_discrete("Kategorien der AV (dep_var_binary)")+
theme_bw()+
ggtitle("Titel der Grafik")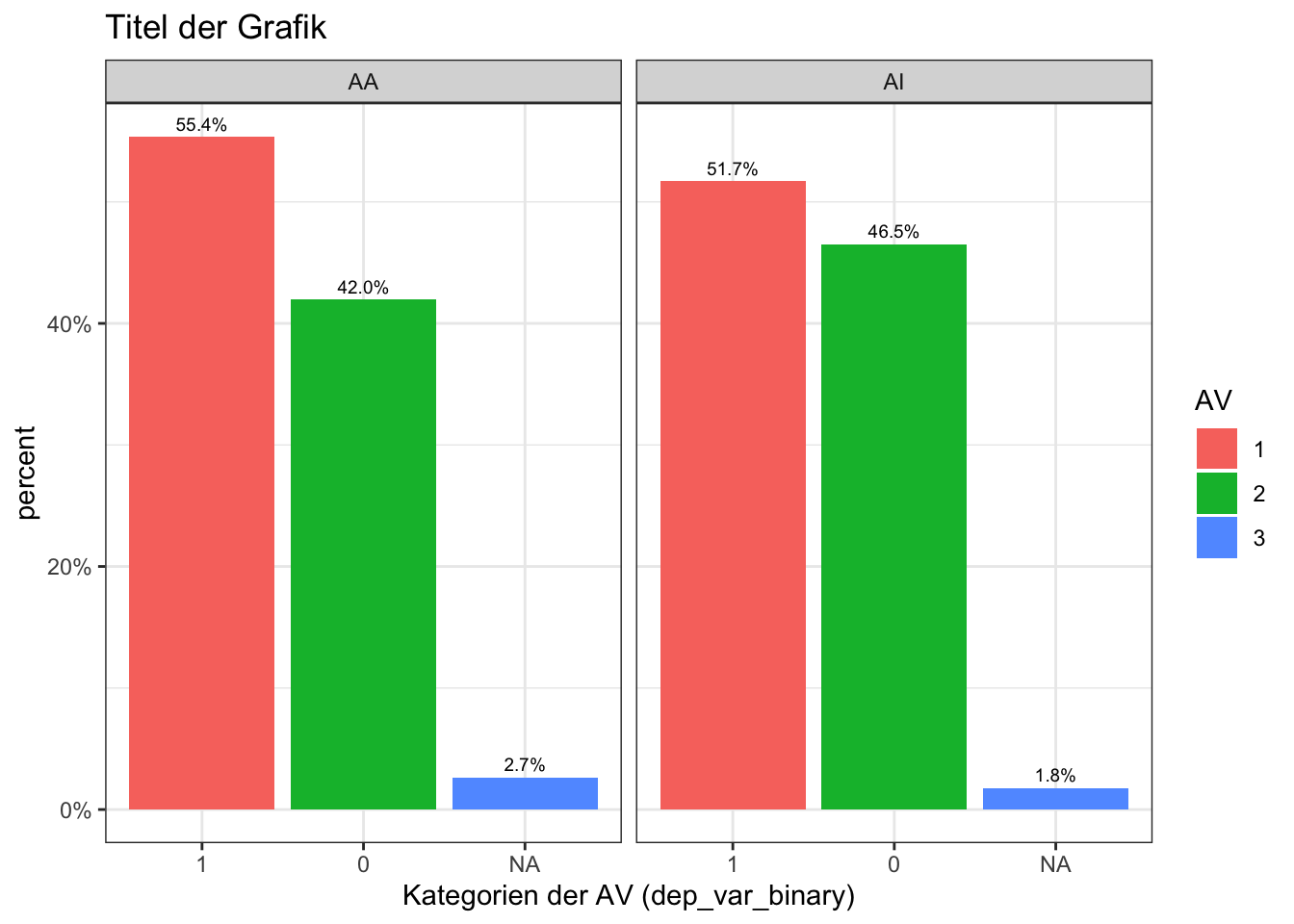
- eigentlich benötigen wir die Legende nicht; wir werden sie ab jetzt ausblenden, und zwar mit “theme(legend.position =”none“)”
- die Farbe der Balken wird im nächsten Beispiel durch die Funktion “scale_fill_brewer” gesteuert; in diesem Fall wählen wir die Farbpalette “Paired” (Infos zu “color brewer”)
- außerdem benennen wir die Kategorien der AV (dep_var_binary) um, mit dem labels-Argument (labels = c(“yes”,“no”,“not available”))
ggplot(data=data, aes(x= dep_var_binary, group=condition2)) +
geom_bar(aes(y = ..prop.., fill = factor(..x..)), stat="count") +
geom_text(aes( label = scales::percent(..prop.., accuracy = 0.1),
y= ..prop.. ), stat= "count", vjust = -.5, size = 2.5) +
labs(y = "percent", fill="AV") +
facet_grid(~ condition2) +
scale_y_continuous(labels = scales::percent)+
scale_x_discrete("Kategorien der AV (dep_var_binary)", labels = c("yes","no","not available"))+
scale_fill_brewer(palette="Paired")+
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")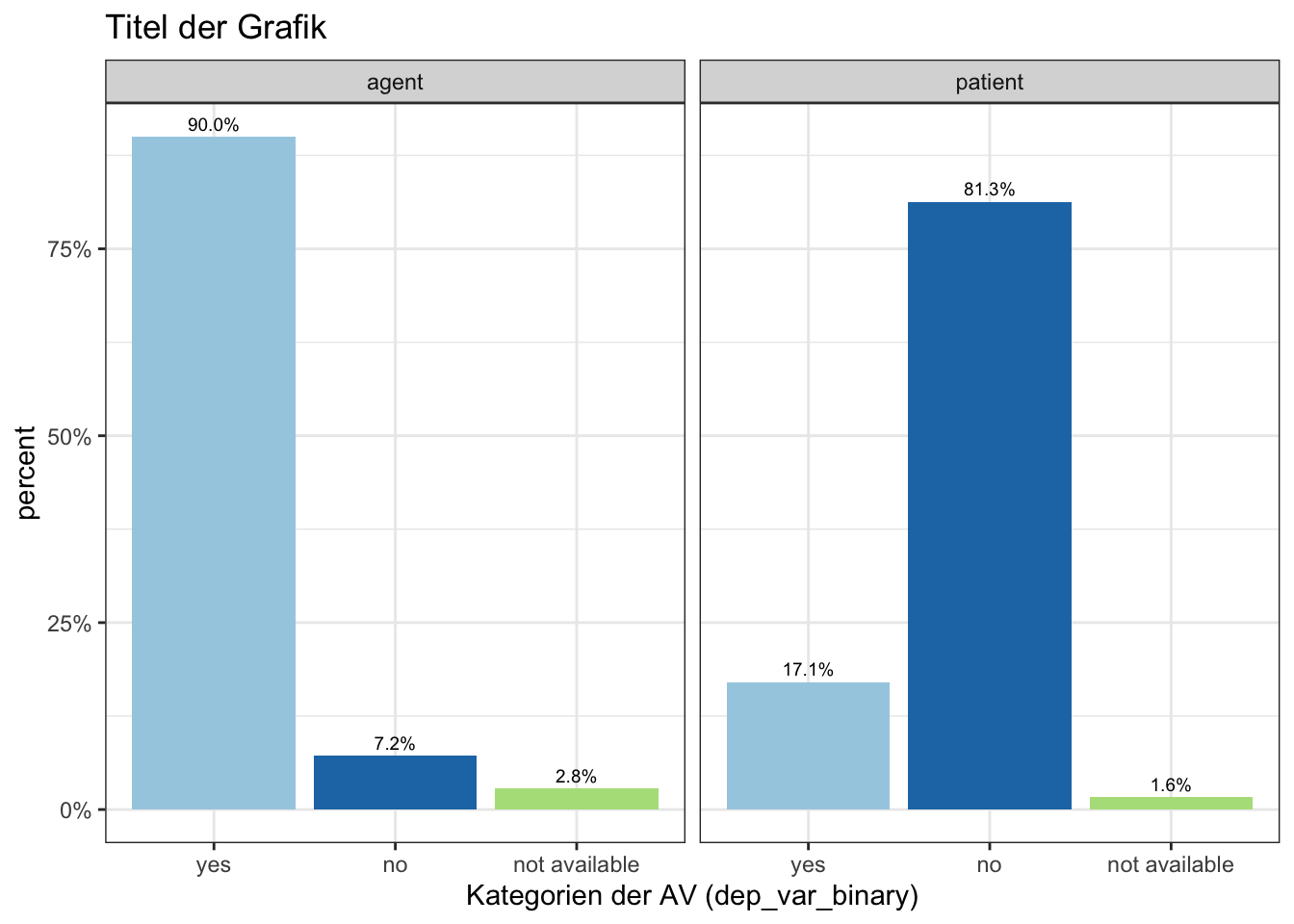
4.1.2 mehrere Variablen
- wir plotten als nächstes die Daten für insgesamt vier Bedingungen (condition1 und condition2 mit jeweils zwei Ausprägungen, 2x2-Design)
- außerdem entfernen wir NA-Werte mit der subset-Funktion
- wir geben der Funktion “scale_fill_brewer” einen neuen Wert: “Accent”
ggplot(data=subset(data,!is.na(dep_var_binary)), aes(x= dep_var_binary, group=condition1)) +
geom_bar(aes(y = ..prop.., fill = factor(..x..)), stat="count") +
geom_text(aes( label = scales::percent(..prop.., accuracy = 0.1),
y= ..prop.. ), stat= "count", vjust = -.5, size = 2.5) +
labs(y = "percent", fill="AV") +
facet_grid(~ condition1 + condition2) +
scale_y_continuous(labels = scales::percent)+
scale_x_discrete("Kategorien der AV (dep_var_binary)")+
scale_fill_brewer(palette="Accent")+
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")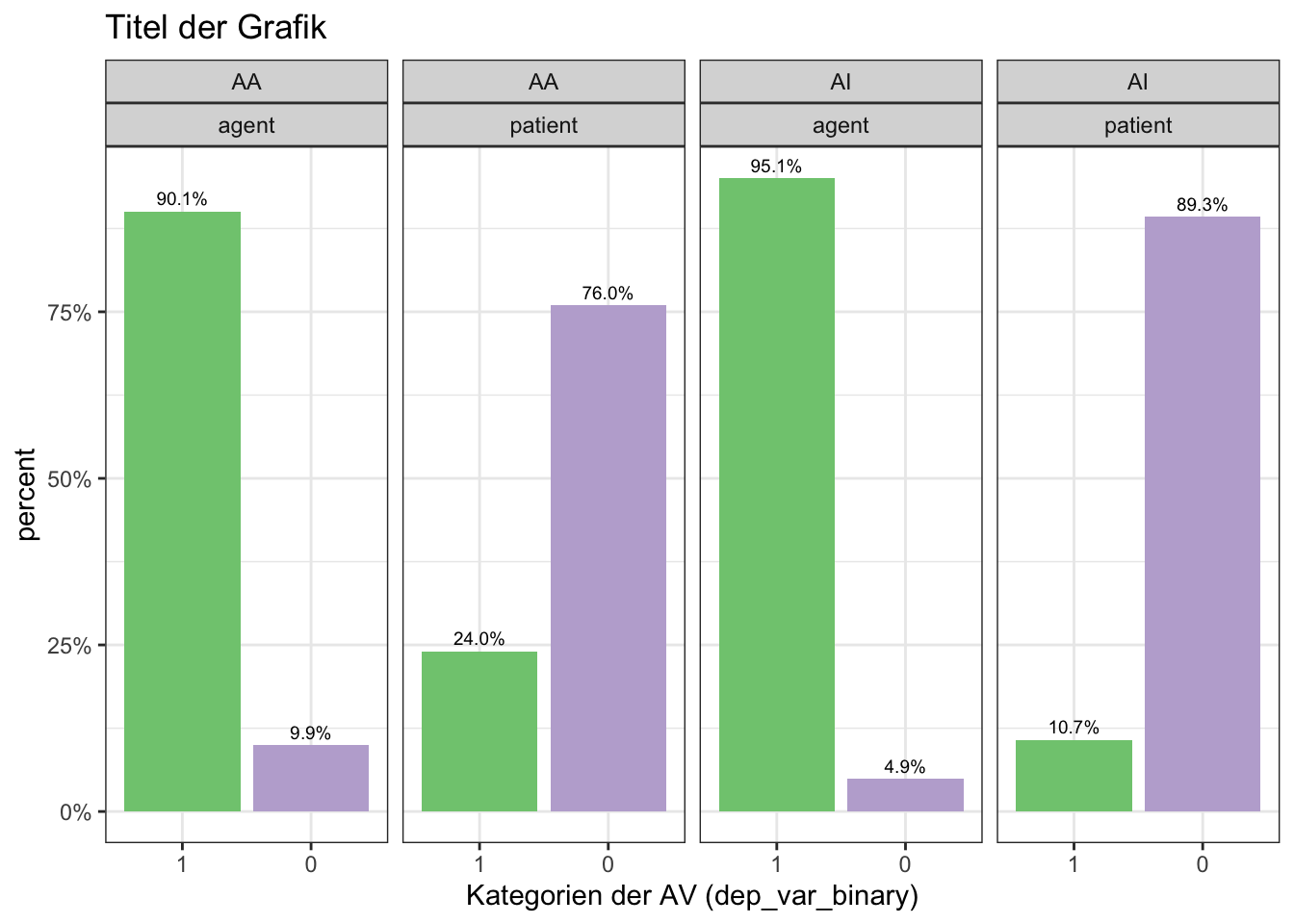
4.2 Mittelwerte
- ich verwende hier und in den nächsten Beispielen die “stat_summary”-Funktion; diese Funktion erlaubt es direkt beim Erstellen der Grafik - also ohne die Rohdaten vorher in irgendeiner Weise “anzufassen” - Mittelwerte und Maße der Varianz darzustellen (mehr Infos hier)
4.2.1 eine Variable
- zuerst stellen wir Mittelwerte und eine Angabe für die Varianz für condition1 dar
ggplot(data, aes(x=condition1, y = dep_V_numeric, color = condition1)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "pointrange") +
scale_color_brewer(palette="Set1")+
labs(y = "gemittelte Reaktionszeit", x="Bedingung (hier: condition1)") +
theme_bw()+
ggtitle("Titel der Grafik")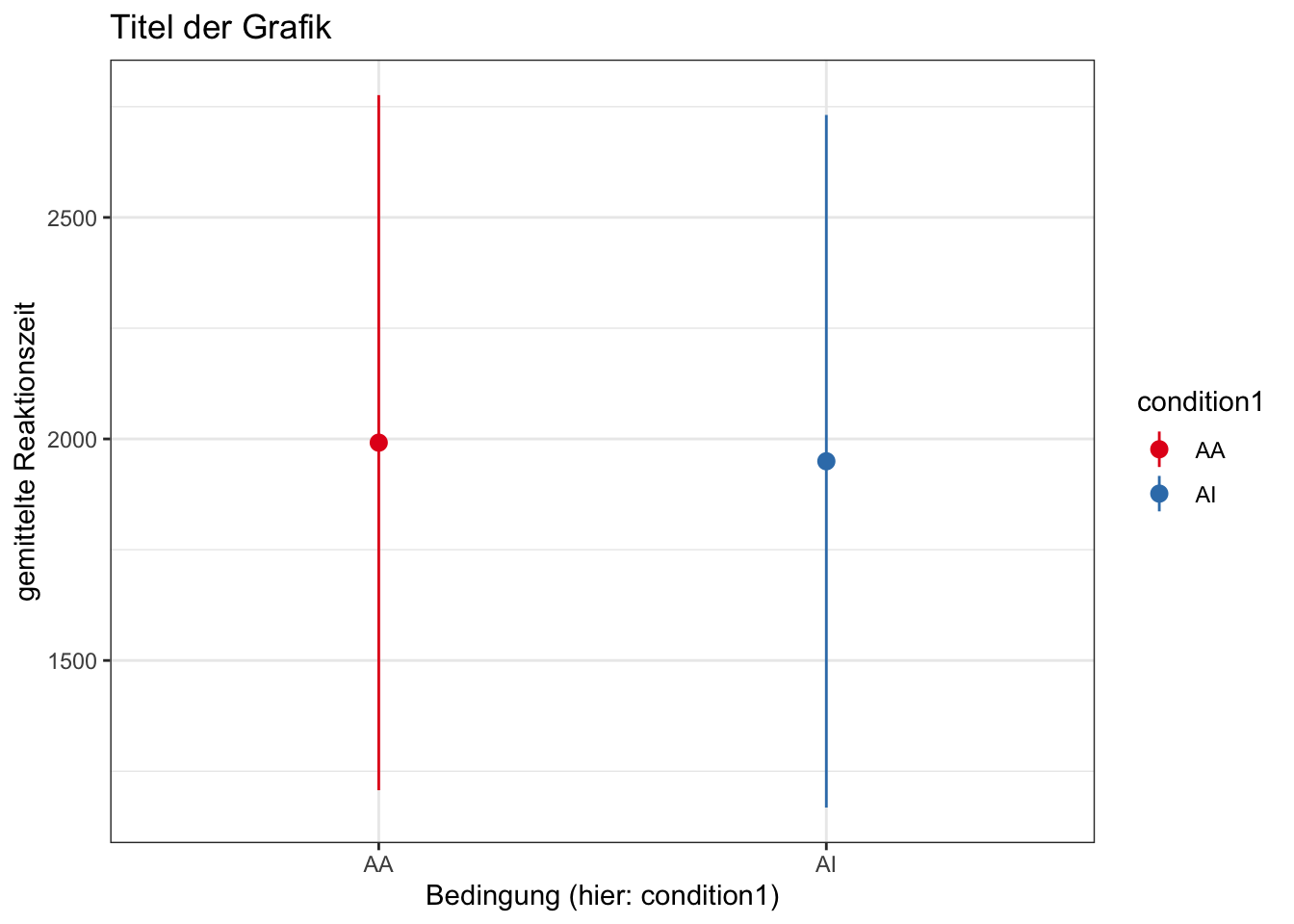
4.2.2 mehrere Variablen
- wie oben, stellen wir als nächstes Mittelwerte und Varianz für mehrere Bedingungen dar
- zur Veranschaulichung wird im nächsten Beispiel innerhalb der “stat_summary”-Funktion ein anderes Maß für die Varianz berechnet: 95%-Konfidenzintervalle
- außerdem wird die Legende wieder verborgen
- durch die Funktion “facet_wrap()” werden die Panels erzeugt (mehr zu “facet_wrap()”)
ggplot(data, aes(x=condition1, y = dep_V_numeric, group = condition2, color = condition1)) +
stat_summary(fun.data ="mean_cl_boot", geom = "pointrange") +
scale_color_brewer(palette="Set1")+
labs(y = "gemittelte Reaktionszeit", x="Bedingung (hier condition2)") +
facet_wrap(~condition2)+
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")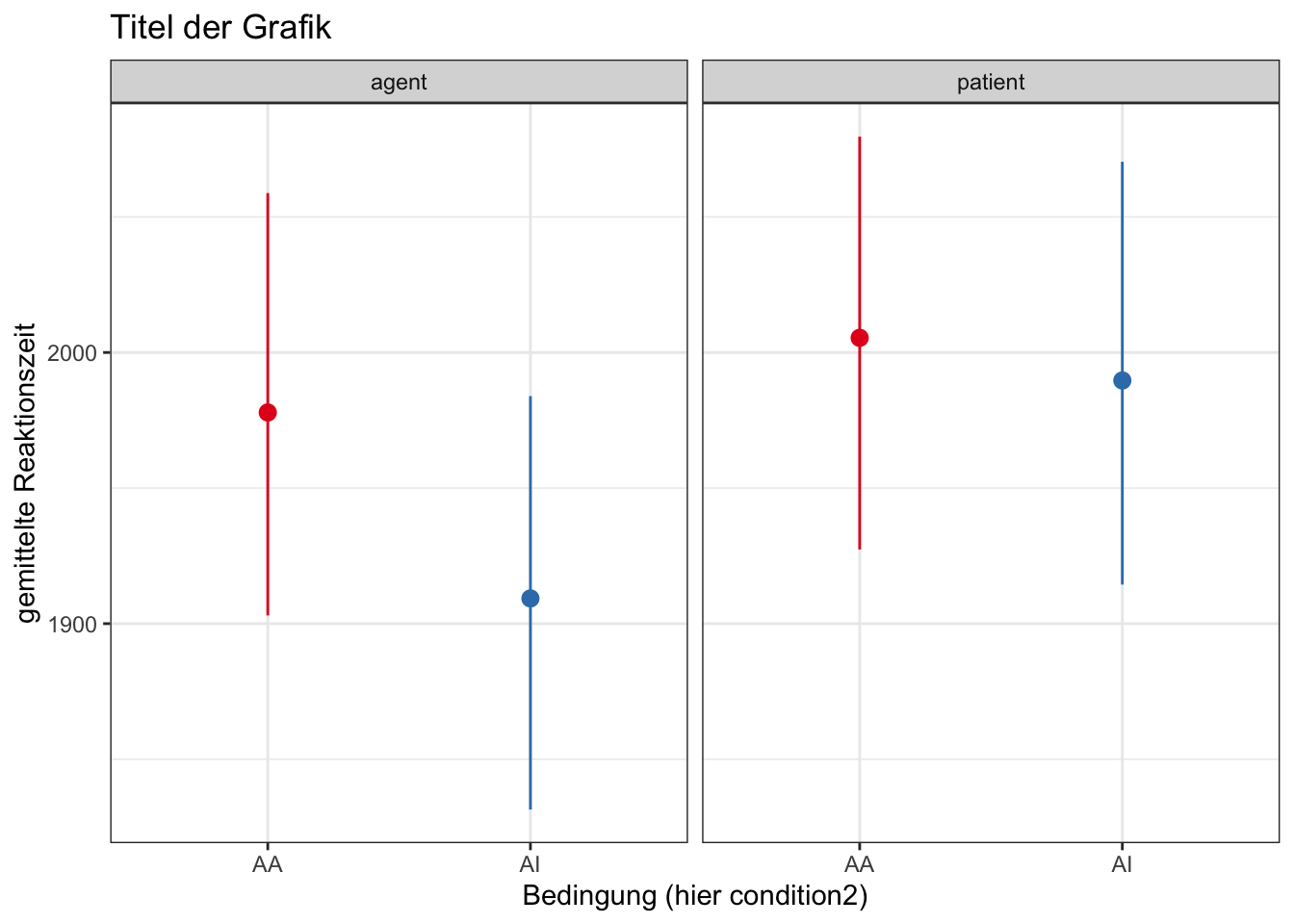
4.3 Zeitreihenanalyse
- in einigen Situationen möchte man den Verlauf der Zeit bei der Datenexploration berücksichtigen; zum Beispiel könnte es interessant sein, zu analysieren, ob sich gemessenene Werte zwischen Anfang und Ende eines Experiments verändern
4.3.1 gesamtes Datenset
ggplot(data, aes(x=trial_ID, y = dep_V_numeric)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5)+
stat_summary(fun ="mean", geom = "line", color = "#8A2BE2", size = 1.5) +
stat_summary(fun ="mean", geom = "point", color = "black", size = 1.5) +
scale_color_brewer(palette="Paired")+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")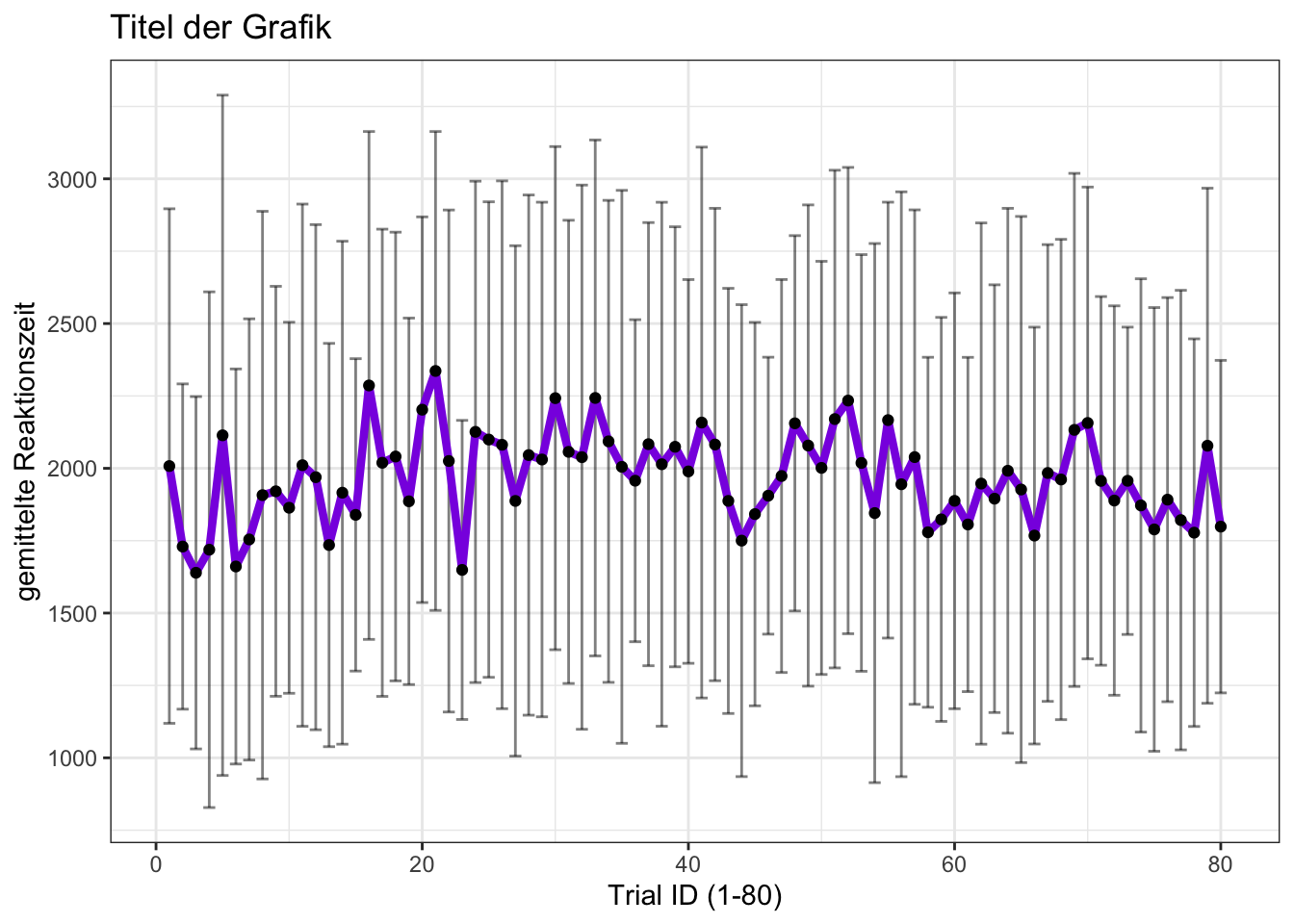
- in dieser Grafik gibt es einiges zu entdecken:
- die schwarzen Punkte geben die Mittelwerte zu jedem Messzeitpunkt an
- die vertikalen Linien geben die Standardabweichung für jeden Mittelwertpunkt an
- um die Punkte, die die Mittelwerte abbilden, miteinander mit einer Linie zu verbinden, wird hier noch eine eigene “stat_summary”-Funktion verwendet
- übrigens hat die Reihenfolge der drei stat_summary-Funktionen einen Einfluss auf die Darstellung: in diesem Beispiel werden zuerst die Standardabweichungen geplottet und auf einem Layer oben drüber, die Linie, und darüber die Punkte; das könnte man auch, je nach Situation, andersherum machen
- zur Veranschaulichung habe ich hier bei der Standardabweichung auch den Parameter “alpha” verwendet; alpha gibt die Deckkraft an; in den folgenden Beispielen verändern wir den Wert für alpha noch ein paar Mal
- die Größe von Punkten und die Dicke von Linien können mit dem “size”-Parameter gesteuert werden
- die Farbe der Linie wird in diesem Beispiel manuell als Hexadezimal-Zahl angegeben (“#8A2BE2”)
- die Farbe der Punkte wird auch manuell angegeben, aber hier als “Wort”-Wert (“black”)
4.3.2 eine Variable
- es gibt mehrere Möglichkeiten, Daten in Abhängigkeit von Bedingungen darzustellen; im nächsten Beispiel geschieht es mit Farben
- in diesem Beispiel verwenden wir für die color_brewer-Funktion die Palette “Set1”
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition1)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.3)+
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Set1")+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")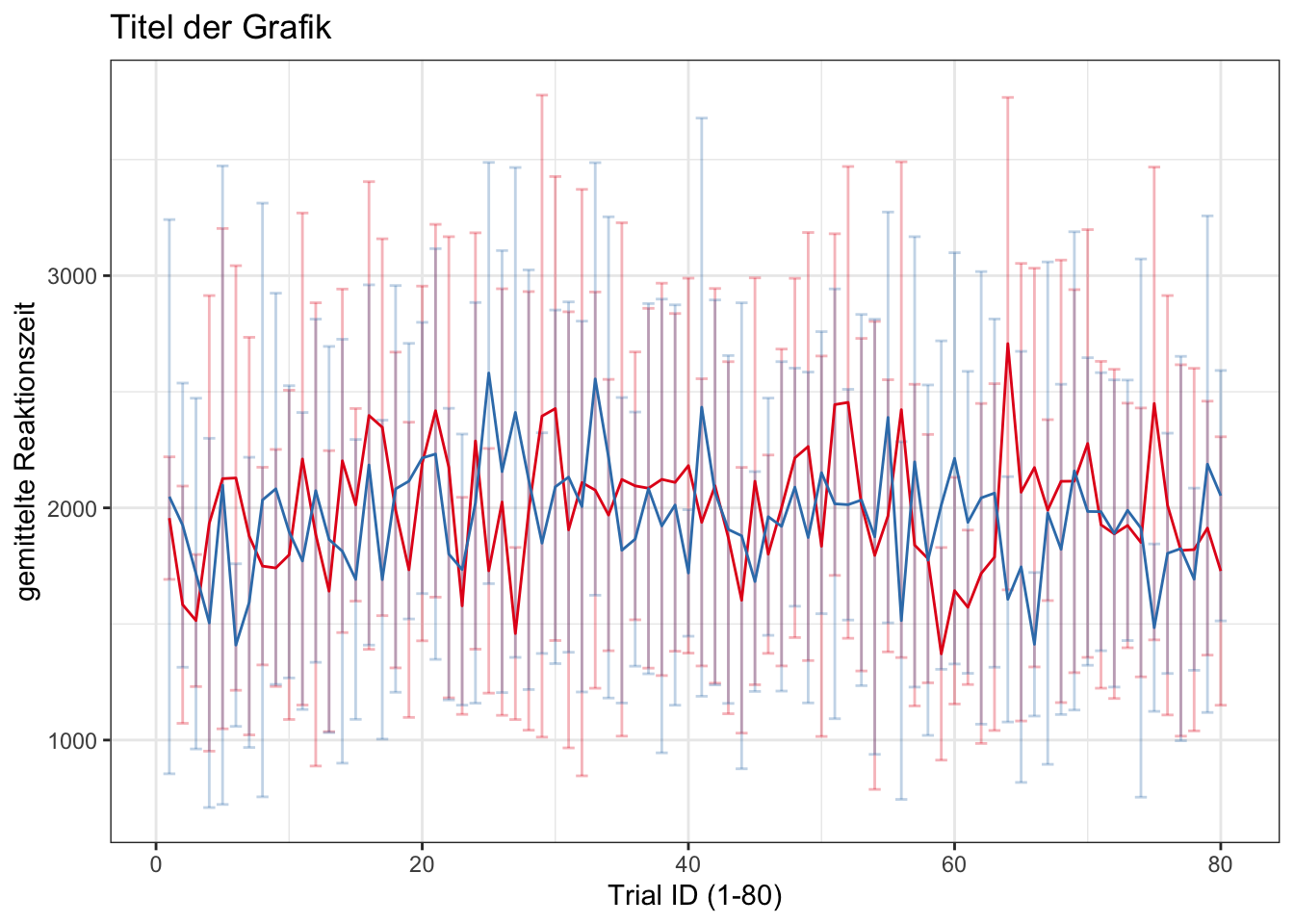
- so richtig gut sieht man aber im Beispiel oben die Unterschiede zwischen den beiden Bedingungen nicht
- wir können beide Bedingungen auch jeweils in einem eigenen Panel darstellen
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition1)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5)+
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Set1")+
facet_wrap(~condition1)+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")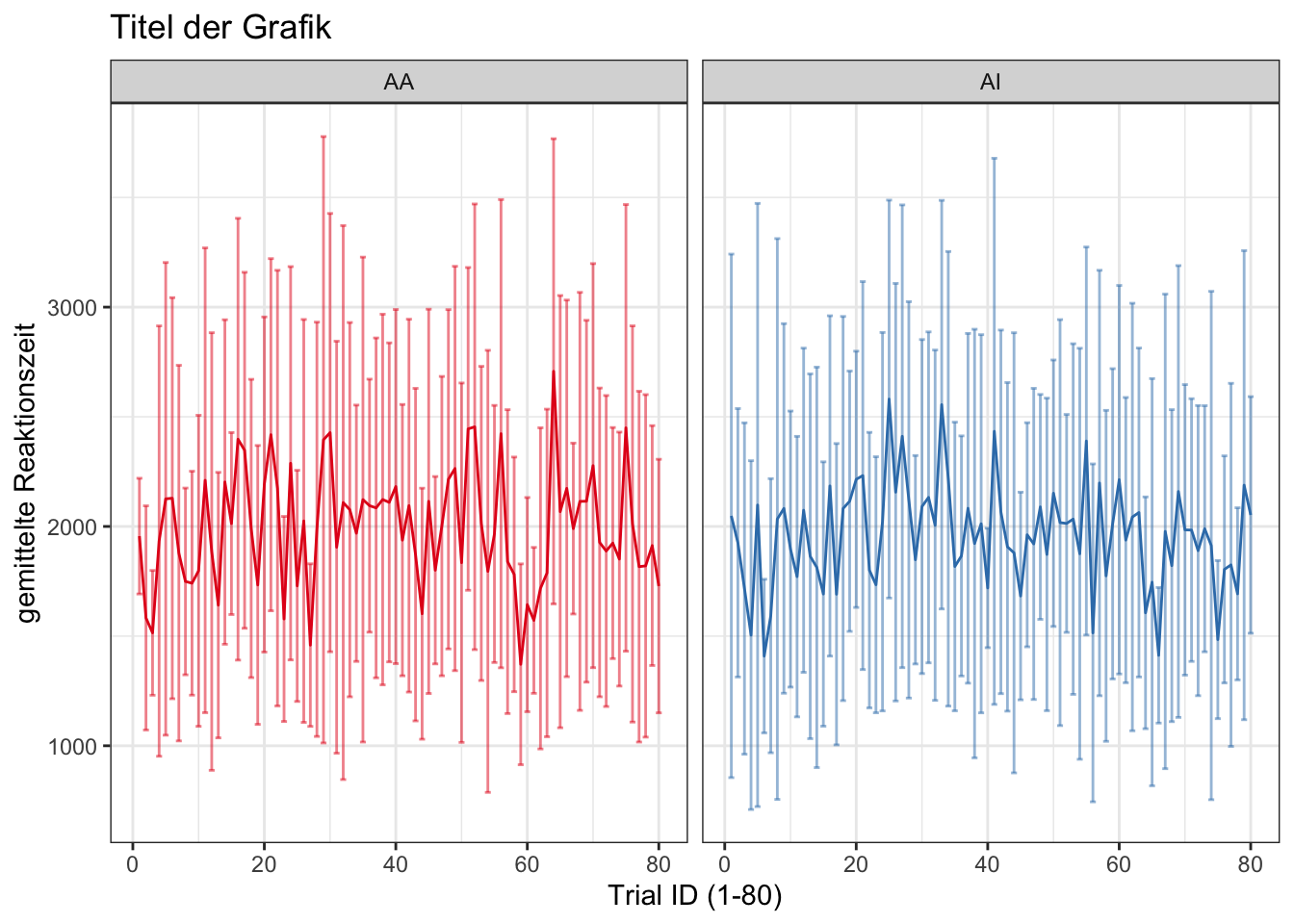
4.3.3 mehrere Variablen
- durch die Verwendung der “facet_wrap”-Funktion ist die Darstellung von mehr als einer Bedingung einfach abzuleiten
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition1)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5)+
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Accent")+
facet_wrap(~condition1+condition2)+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")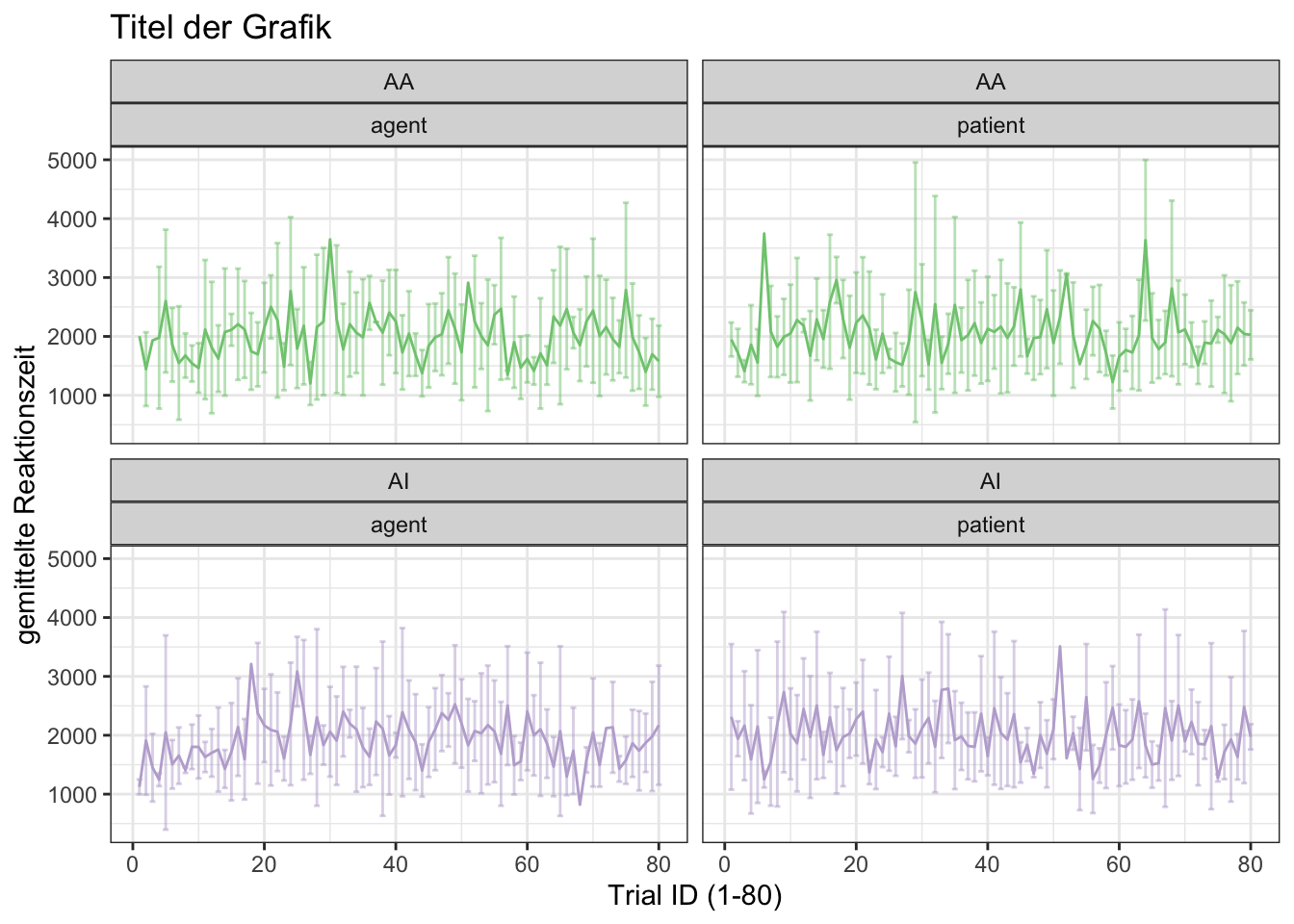
- wir können die Darstellung der Panels auch anpassen; im nächsten Beispiel gibt “ncol” an, dass die Panels in nur einer Spalte (engl. column; “ncol” steht für “number of columns”) dargestellt werden sollen
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition1)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5)+
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Accent")+
facet_wrap(~condition1+condition2, ncol = 1)+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
theme(legend.position = "none") +
ggtitle("Titel der Grafik")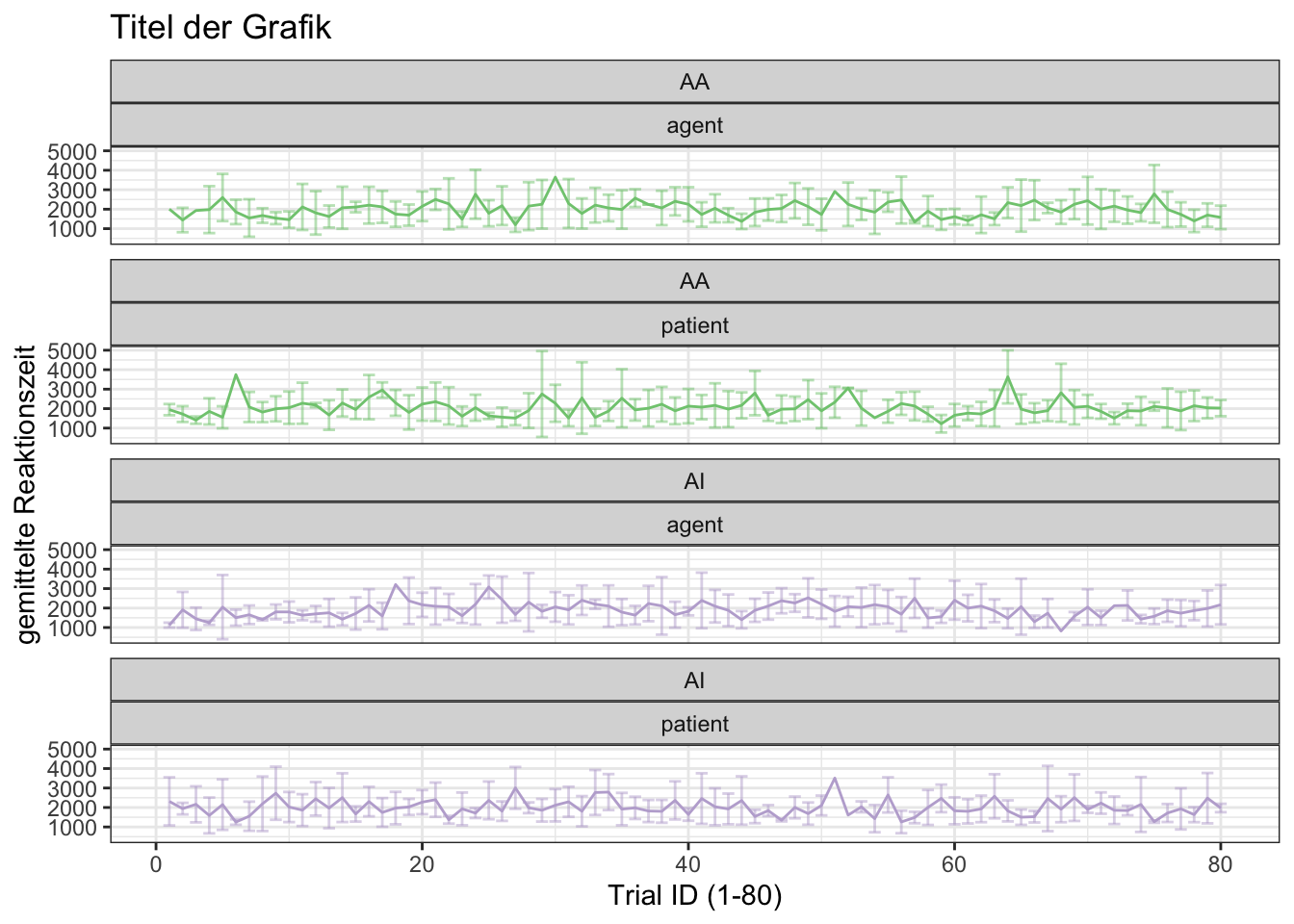
- im vorletzten Beispiel werden die Panels in 4 Spalten dargestellt und die Farbkodierung wird durch condition2 bestimmt
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition2)) +
stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5)+
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Accent")+
facet_wrap(~condition1+condition2, ncol = 4)+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
ggtitle("Titel der Grafik")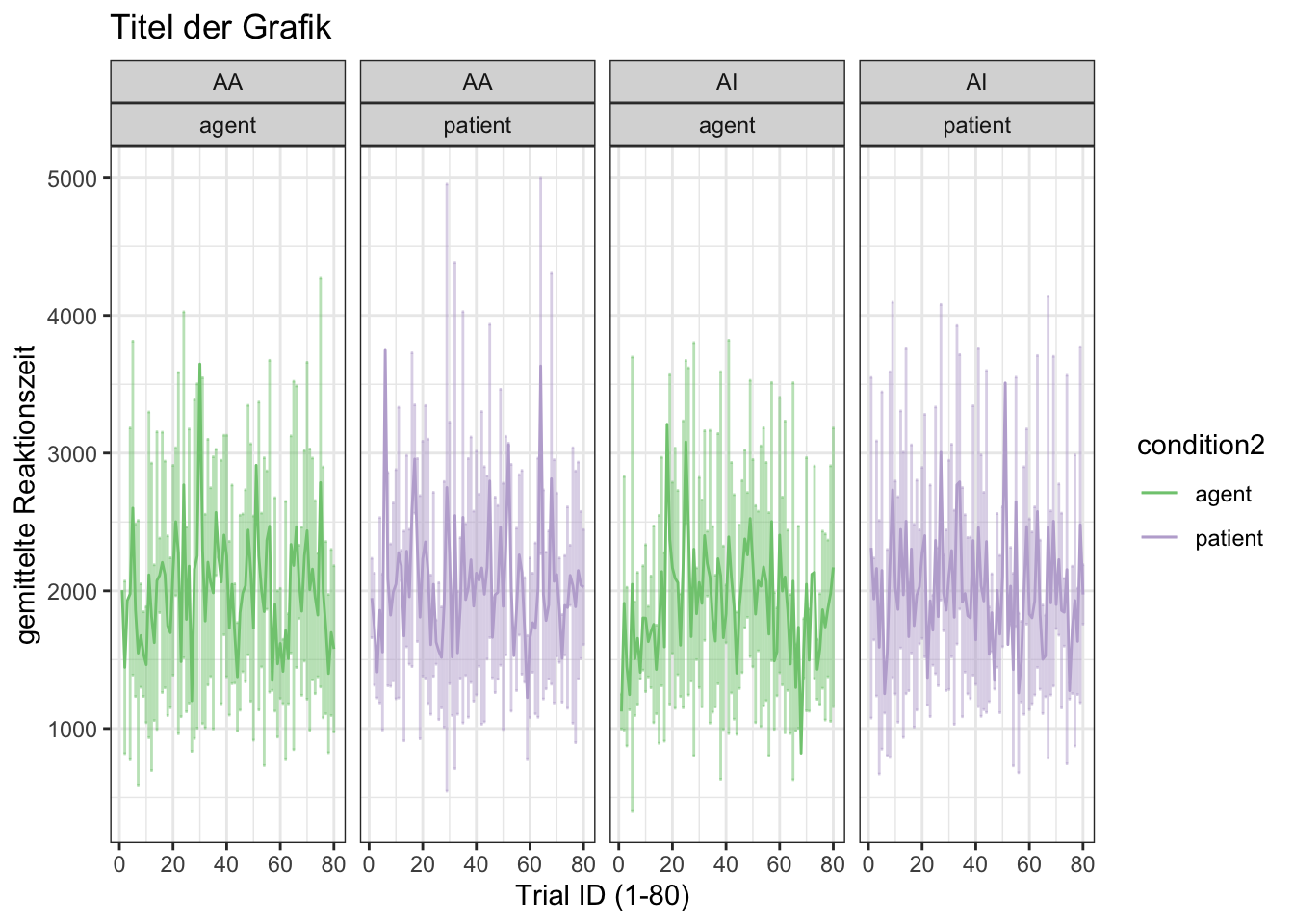
- in zwei Spalten ist es wohl am besten; jetzt lassen wir die Standardabweichung weg; dafür benutzen wir ausnahmsweise die Kommentar-Markierung “#” (Code mit “#” wird von R nicht interpretiert)
ggplot(data, aes(x=trial_ID, y = dep_V_numeric, color = condition2)) +
#stat_summary(fun.data ="mean_sdl",fun.args = list(mult = 1), geom = "errorbar", alpha = 0.5) +
stat_summary(fun ="mean", geom = "line") +
scale_color_brewer(palette="Dark2")+
facet_wrap(~condition1+condition2, ncol = 2)+
labs(y = "gemittelte Reaktionszeit", x="Trial ID (1-80)") +
theme_bw()+
ggtitle("Titel der Grafik")+
theme(legend.title=element_blank())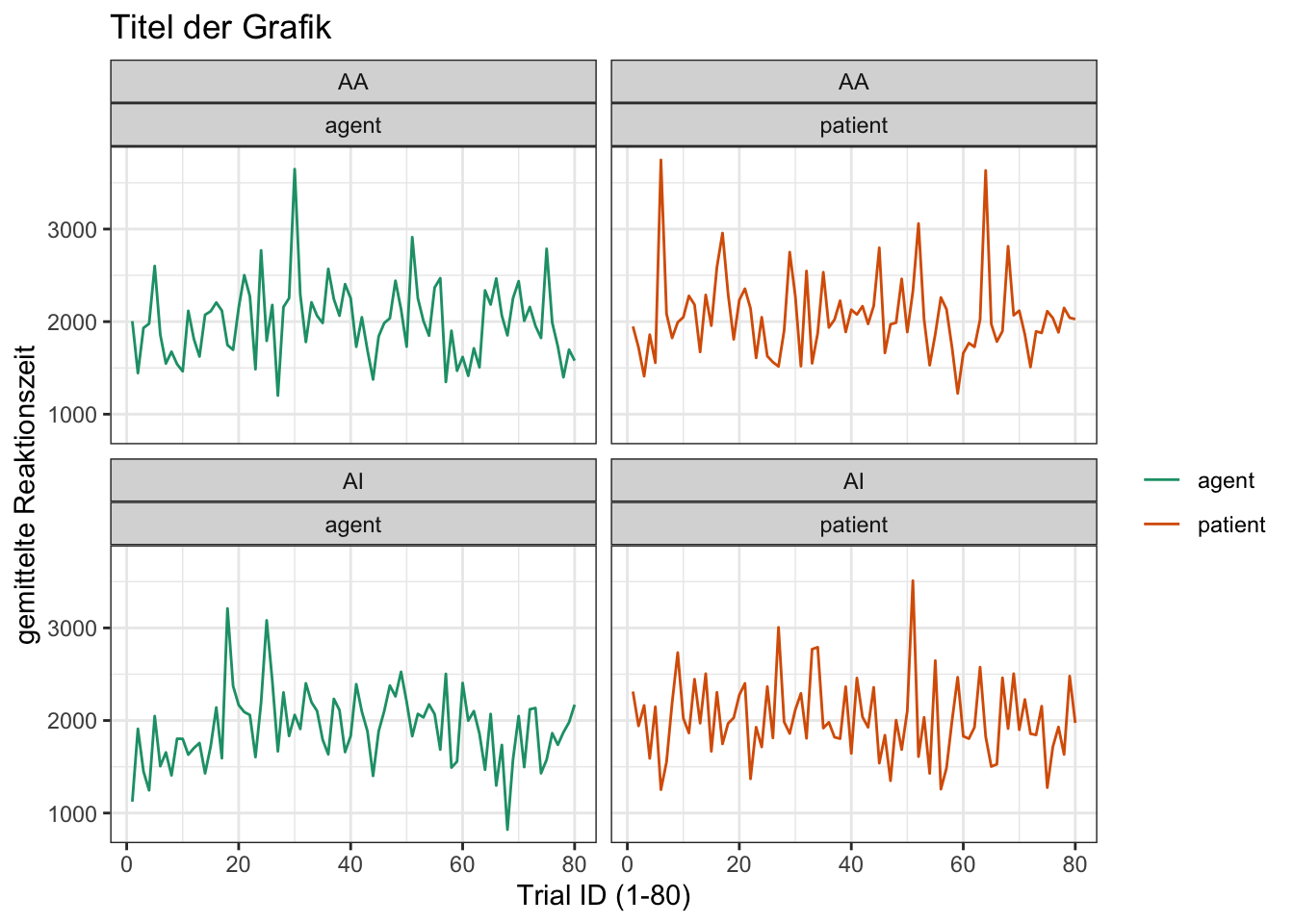
5 Grafiken speichern
- Mit ggsave() kann man erstellte Grafiken in einem bestimmten Format, in einer bestimmten Größe, an einem betsimmten Ort abspeichern
- ggsave ist auch Teil des packages “ggplot2”
- Name und Ort der Datei:
- Im folgenden Beispiel wird eine jpg-Datei mit dem Namen “Dateiname.jpg” im Unterordner “Plots” gespeichert; ohne “./plots/” würde die Datei direkt im R Projekt-Ordner gespeichert werden
- Größe und Auflösung:
- im Beispiel unten wird die Grafik in der Größe 8 inch mal 4 inch mit einer Auflösung von 300 dpi generiert (Es empfiehlt sich an den Parametern für Größe etwas rumzuspielen bis das Ergebns zufriedenstellend ist)
ggsave(filename = "./plots/Dateiname.jpg", last_plot(), width =8, height =4, dpi =300)